5. Genetik: Besonderheiten der Bakteriengenetik
5.1. Struktur der DNA
Grundsätzliche Kenntnisse über die molekulare Struktur
der Nukleinsäure werden für dieses Kapitel vorausgesetzt. An dieser Stelle wird
lediglich ein Kurzüberblick gegeben.
Der
Träger der genetischen Information, die DNA (für engl. Deoxyribonucleic
acid) ist in ihrer Grundstruktur bei allen Organismen
sowie den Viren weitgehend identisch. Vier Basen repräsentieren den genetischen
Code wobei jeweils drei Basen die Information für ein Triplett
darstellen; ein Triplett codiert für eine Aminosäure.
Die Basen liegen in Form von Nukleotiden vor, welche
in spezifischer Weise verknüpft eine DNA-Doppelhelix
ergeben. Innerhalb dieser Doppelhelix kommt es über
Wasserstoffbrückenbindungen zu komplementären Basenpaarungen. Biologischer Sinn
der Doppelhelix ist die Möglichkeit der
semikonservativen Reduplikation, die es ermöglicht, das bei der Zellteilung
beide Tochterzellen eine (zumindest theoretisch) identische Ausstattung an
Erbgut erhalten. Mutationen, die in
gewissen Abständen die DNA-Sequenz verändern, führen zu genetischer
Variation und
werden damit zum Motor für Anpassung, Adaptation und Evolution. Mutationen
können ohne Folgen bleiben (z.B. weil eine Mutation die dritte Position in
einem Triplett betrifft und damit mit hoher
Wahrscheinlichkeit nicht zu einer Aminosäuresubstitution führt), Mutationen
können zum Nachteil des Individuum sein (z.B. letale Mutationen oder solche,
welche die Fitness des Individuums reduzieren), oder sie können —entweder
sofort oder bei einem Wechsel der Umweltbedingungen— dem Individuum einen
Vorteil bringen (à siehe Antibiotikaresistenzen).
Die
wesentlichen Unterschiede bezüglich des Erbgutes liegen bei Pro- bzw. Eucaryonten in der Organisation des Erbgutes. Neben
nachstehenden Informationen zum grundsätzlichen Aufbau der DNA sind die
weiteren Unterschiede in à Tabelle 6 (S. 29)
zusammengefasst:
Ø
Eucaryonten: Die DNA wird durch Histone
(Histonproteine) stabilisiert. Diese Histone liegen in regelmäßigem Abstand vor. Der
solchermaßen stabilisierte „DNA-Faden“ wird mehrfach spiralisiert
so dass eine Kondensierung zu mikroskopisch sichtbaren Chromosomen erfolgt.
Ø
Procaryonten: Keine Chromosomen sondern Chromosomenäquivalent, d.h.
der „DNA-Faden“ liegt als superspiralisierte Struktur
vor. Es gibt weder Histone noch eine Bildung von Chromatin.
5.2.
Unterschiede
zwischen der Eucaryonten- und Procaryontengenetik
Tabelle 7: Wesentliche
Unterschiede zwischen den Pro- und Eucaryonten im
Hinblick auf die Genetik:
|
Struktur |
Procaryonten |
Eucaryonten |
|
Chromosomen |
Chromosomen-Äquivalent,
ringförmige DNA |
echte Chromosomen |
|
immer haploid |
(haploide), diploid (tetraploid oder polyploid) |
|
|
Vorkommen von Plasmiden |
keine Plasmide |
|
|
Genomgröße |
2-4 x 106 |
109 und mehr |
|
DNA Struktur |
kein höherer
Ordnungszustand |
DNA auf Histonproteine aufgespult und superspiralisiert |
|
genetische Regulation |
Gen als funktionelle
Einheit |
Gene oft aus Introns und Exons aufgebaut |
|
|
räumliche Trennung von
Transkription und Translation |
|
|
polycistronische mRNA |
monocistronische mRNA |
|
|
kein splicing |
splicing der RNA |
Neben
den unten angesprochenen Unterschieden auf Ebene der Regulation der DNA bzw.
genetischer Vorgänge bestehen noch weitere Unterschiede betreffend der Ribosomen und den an genetischen Prozessen beteiligten
Enzyme. Ribosomen und die RNA-Polymerase
stellen unabkömmliche, essentielle Bestandteile jeder Zelle dar, da sie eine
zentrale Rolle in der Proteinbiosynthese spielen. Die Grundstruktur der beiden
Komponenten ist jedoch unterschiedlich, wenn man Eu- und Procayronten
vergleicht. Diese Strukturunterschiede erlauben es, die
bakteriellen Strukturen gezielt durch Antibiotika zu hemmen ohne die
entsprechenden eucaryontischen Strukturen in ihrer
Funktion zu beeinträchtigen (verg. aber à Mutationen à Antibiotika-Resistenzen und à genetische Variabilität).
Eine
gute Abhandlung über die strukturellen Besonderheiten der bakteriellen RNA-Polymerase und die Wirkung des Rifampicin
gibt: Campbell et al (2001): Structural Mechanism for Rifampicin Inhibition of Bacterial RNA Polymerase. Cell 104:901-912.
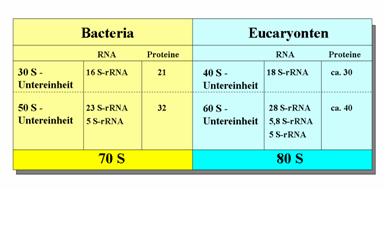
Abb. 17: Unterschiede in der
Struktur der Ribosomen von Bakterien (70S Ribosomen) und Eucaryonten (80S Ribosomen) im Hinblick auf die Ultrastruktur (Proteine, rRNA’s)
5.3.
Genetische
Regulation
5.3.1.
Struktur der Gene
und Transkription:
5.3.1.1.
Eucaryonten:
Ø
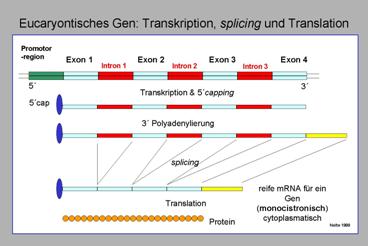
Gene bestehen aus
Introns und Exons als
alternierende subgenische Abschnitte (Abb. 18). Die Exons beinhalten die genetische Information für das
Protein, die Introns sind nicht codierend und werden
nachfolgend durch das à splicing aus der mRNA
entfernt.
Ø
splicing der mRNA: Entfernen der Introns und Anhängen eines sogenannten
poly-A tails à reife mRNA
Ø
monocistronische mRNA`s, d.h., eine mRNA codiert für ein einziges Protein.
Ø
Trennung von
Transkription, splicing (im Zellkern) und Translation (im Cytoplasma)
Abb. 18: Organisation,
Transkription und Reifung von eucaryontischen Genen
bzw. mRNA`s. Nicht dargestellt sind mögliche posttranslationale Ereignisse auf Proteinniveau.
5.3.1.2.
Procaryonten
Ø
keine Unteilung
der Gene in Introns und Exons
(Kolinearität der Gene und Proteine, Abb. 19).
Ø
keine weitere
Reifung der mRNA, diese wird sofort der Translation (Proteinsynthese) zugeführt.
Ø
procaryontische mRNA`s können polycistronisch sein, d.h. es werden mehrere, oft zu einer
strukturellen Einheit gehörende Gene (à Operons) in einer mRNA zusammengefasst.
Ø
keine Trennung
von Transkription und Translation: schon während der
Transkription beginnen Ribosomen an der noch
„unfertigen“ RNA mit der Translation.
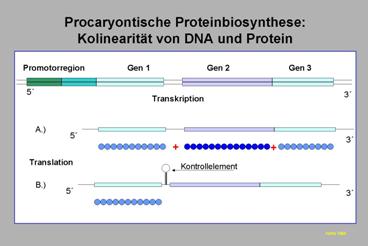
Abb. 19: Organisation und
Transkription von procaryontischen Genen bzw. mRNA`s. Nicht dargestellt sind mögliche posttranslationale
Ereignisse auf Proteinniveau.
5.3.2.
Operons
(intelligent gesteuerte genetische Einheiten)
Mit
Hilfe von Operons sind Bakterien in der Lage,
schnell, flexibel und energetisch günstig ihren Stoffwechsel an neue
Milieubedingungen zu adaptieren. Operons sind oft Substrat-kontrolliert, die Genprodukte kommen erst zur
Expression, wenn das Substrat welches abgebaut werden soll, eine bestimmte
Konzentration in der Zelle erreicht. Beispiel:
lac-Operon
Das lac-Operon oder Laktose-Operon ist
das Beispiel einer Kassette von Genen, deren Genprodukte eine funktionelle
Einheit bilden (Zusammenwirken unterschiedlichster Proteine beim Stoffwechsel
der Laktose: spezifisches Transportprotein = Permease,
Transacetylase und ß-Galactosidase)
und einer intelligenten Regulation durch das zu Zielsubstrat unterliegen. Der lac-Repressor,
der das Operon in Abwesenheit von Laktose negativ
reguliert, wird bei Ansteigen des intrazellulären Laktosespiegels durch Bindung
der Laktose in seiner Konformation sterisch verändert und gibt das Operon
so lange frei, bis die Laktose im Milieu wieder limitierend wird. Durch
anklicken des folgenden link kann eine PowerPoint Animation zur Funktion des
lac-Operons aufgerufen werden: PowerPoint2003
Animation/PowerPoint Version95
und höher Animation).
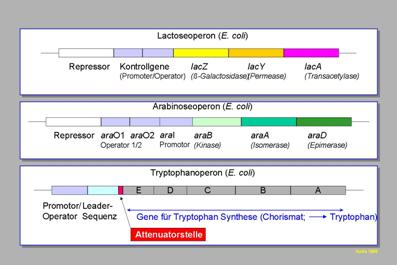
Abb. 20: Struktur bekannter Operons: ara-Operon (Arabinose-Stoffwechsel) his-Operon (Histidin-Biosynthese)
trp-Operon
(Tryptophan-Biosynthese)
Allen
genannten Operons ist gemeinsam, dass sie einer
Regulation durch das Substrat unterliegen. Der genetische Mechanismus variiert
dabei, das Grundprinzip ist aber vergleichbar.
Im
Routinelabor nutzt man die unterschiedlichen
Fähigkeiten der Bakterien bestimmte Kohlenhydrate oder andere Substanzen zu verstoffwechseln (à Stoffwechsel), um
aus Untersuchungsmaterial angezüchtete Bakterien zu
differenzieren (zu identifizieren). Bakterienarten haben i.d.R.
ein für ihre jeweilige Art spezifisches Stoffwechselprofil, welches man mit
Identifikationssystemen abprüfen kann. In Verbindung mit der äußeren Form der
Bakterien und dem Färbeverhalten (à Zellwand) kann
so eine Bakterienart identifiziert werden (siehe auch Kap. 1.2).
5.4.
Genetische Rekombination ...
... oder: „Bakterien haben
Geschlechtsverkehr“!
Sexualität
ist im Grunde nichts anderes als der Austausch und die Rekombination
von Erbgut. Diese Vorgänge finden auch bei Bakterien statt und sorgen neben den
Mutationen für eine stetige Veränderung und Durchmischung (Rekombination) des Erbgutes und damit für eine stetige
Anpassung an sich ändernde Lebensräume. Dabei geschieht die Veränderung des
genetischen Materials der Population (sogenannter gene pool) stetig
und unabhängig von tatsächlich stattfinden Umweltbedingungen. Vielmehr wird die
entstehende genetische Variabilität unter sich tatsächlich verändernden
Umweltbedingungen bedeutsam (à genetische Variabilität).
5.4.1.
Konjugation
Austausch von Plasmiden
(kurze, ringförmige DNA`s mit zusätzlicher
genetischer Ausstattung wie z.B. Resistenzgene). Plasmide können innerhalb einer Population einer bestimmten
Bakterienart weitergegeben werden oder sogar zwischen verschiedenen
gram-negativen Bakterien. Modellcharakter hat der sogenannte
F-Pilus (auch Sex-Pilus)
bei E. coli.
Über diesen F-Pilus übertragen F+-Bakterien
das Plasmid auf F- Empfängerbakterien.
5.4.2.
Transduktion
Veränderung der Bakterien durch Bacteriophagen,
diese transferieren entweder bakterielle DNA zwischen den Bakterien (klassische
Transduktion) oder verändern Bakterien durch Einbau
zusätzlicher Phagen-kodierter Gene (Lysogenisierung z.B. von Corynebacterium diphtherie
(wird dann zum Toxinbildner) oder Streptococcus pyogenes (wird ebenfalls zum
Toxinbildner)).
5.4.3.
Transformation
ungerichtete Aufnahme freier DNA aus dem Milieu (DNA lysierter Mikroorganismen kann von einigen Bakterien wie Neisseria-Arten
[u.a. Erreger der Gehirnhautentzündung und der Gonorrhoeae] aufgenommen werden). Hierbei treten unter
Umständen Rekombinationsereignisse ein, welche die
genetische Variabilität der Bakterien erhöhen.
Die
beschriebenen Vorgänge dienen dem Austausch und der damit verbundenen Rekombination von Erbgut. Rekombination
führt zur Erhöhung genetischer Variabilität. Genetische Variabilität ist
Grundlage der Adaptation an wechselnde Umweltbedingungen und damit Motor der
Evolution.
Frage zur eigenen Auseinandersetzung mit
dem Stoff:
Überlegen Sie sich die praktische Relevanz welche die Weitergabe von
Resistenzfaktoren über Plasmide in der Umwelt hat.
5.5.
Vorteile
genetischer Variabilität — Anpassung an das Milieu
Genetische
Einheitlichkeit stellt eine Sackgasse dar. Nur Populationen, die eine gewisse
genetische Variabilität aufweisen können, sind in der Lage sich auf wechselnde
Umweltbedingungen einzustellen (genetische
Variabilität ist eine Eigenschaft von Populationen, nicht von Individuen,
Abb. 21). Die mit genetischer Variabilität verbundene Anpassungsfähigkeit kann eine Eigenschaft der Population sein
(unter optimalen Umweltbedingungen hält der gene pool der Population genügend
Variabilität für eventuell wechselnde Umweltbedingungen vor). Bei schnellen und
extremen Veränderungen in der Umwelt oder bei Besiedlung neuer Lebensräume
stellt Anpassungsfähigkeit eine Individuelle Eigenschaft dar. Der unter
veränderten Umweltbedingungen geeignete Ausschnitt aus dem gesamten gene pool sichert
das weitere Überleben der Art bzw. ermöglicht die Konstanz der von den
veränderten Umweltbedingungen betroffenen Population. Die sich mit diesen
Fragen befassende Disziplin der Naturwissenschaften ist die Populationsgenetik.
Populationsgenetische Vorgänge haben weit mehr als nur theoretische Bedeutung.
Jede Besiedlung und jede Infektion des Menschen wie auch jeder lenkende
Eingriff durch Antibiotika stellt einen Eingriff in die Populationsgenetik und
–dynamik der betreffenden Bakterienpopulation dar.
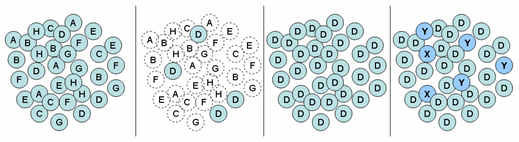
|
Abb. 21: Beispiel für eine
Bakterienpopulation unter optimalen Umweltbedingungen. Durch genetische Rekombination und Mutation sind insgesamt 8 (A-H)
unterschiedliche Varianten in der Population vertreten. Alle Varianten haben
unter den normalen Bedingungen eine vergleichbare Überlebenswahrscheinlichkeit. |
|
Ein Wechsel der Umweltbedingungen führt zum
Erlöschen der nicht an die geänderten Bedingungen angepassten Individuen.
Varianten, deren individuelle Eigenschaft zu den neuen Umweltbedingungen
passt, sichern die Arterhaltung und das weitere Bestehen der Population. Über
die Zeit wird neue genetische Variabilität entstehen. |
Die Folgen
dieser Betrachtung werden am Beispiel der Antibiotikaresistenzen deutlich.
In einer den Menschen besiedelnden Population von Bakterien, in der zufällig
eines der Bakterien durch eine Punktmutation die Eigenschaft erwirbt, resistent
gegen Rifampicin (first line Antibiotikum gegen den Erreger der
Tuberkulose, M. tuberculosis)
zu sein (z.B. Variante D in Abb. 21), sind zunächst einmal alle Bakterien mehr
oder weniger vergleichbar gut an den aktuellen Lebensraum angepasst. Verändert
sich das Milieu im Sinne eines ansteigenden Konzentration auf Grund einer
Behandlung mit Rifampicin haben die durch Teilung
entstandenen Tochterbakterien des resistent gewordenen Bakteriums einen
erheblichen Standortvorteil und sind in der Lage, den Fortbestand der
Population zu sichern. Während also der Großteil der Population auf Grund
Unangepasstheit an die veränderten Umweltbedingungen ausgemerzt wird (Varianten
A-C und E-H in Abb. 21), sichert eine Minderheit, die einen bestimmten
Ausschnitt aus dem gene pool
darstellt, das Überleben der Art. Man spricht von Selektion der am besten
angepassten Variante. Vergleichbare Vorgänge lassen sich in vielen Fällen
beobachten, wenn Antibiotika z.B. nicht in ausreichender Konzentration oder
über einen ausreichend langen Zeitraum gegeben werden.
Von
erheblicher Bedeutung ist, dass zumindest in einigen Fällen der Erwerb der
Eigenschaft Antibiotika-Resistenz dem betreffenden
Träger dieser Eigenschaft unter normalen Umweltbedingungen auch einen
Standortnachteil verschaffen kann. So haben z.B. die Erreger der eitrigen
Meningitis, N. meningitidis, unter
Therapie mit Rifampicin einen eindeutigen Vorteil,
wenn die Mutation zur Rifampicin-Resistenz vorhanden
ist. Unter normalen Umweltbedingungen jedoch kehrt sich dieser Vorteil in einen
Nachteil um. Die Resistenz-vermittelnde Mutation
findet im Gen für die RNA-Polymerase statt. Durch
diese Mutation wird zwar die Resistenz vermittelt, offenbar wird jedoch die Polymerase in ihrer Aktivität langsamer. Dadurch verlängert
sich die Generationszeit der resistenten Erreger. Diese Veränderung ist zwar
nur geringfügiger Natur, reicht jedoch aus, um im Rifampicin-freien
Milieu dem Wildtyp, sprich empfindlichen Stamm unterlegen zu sein. Bringt man
einen resistenten und einen empfindlichen Stamm von N. meningitidis gleichzeitig in eine Kultur ein, so kann man mit
entsprechenden Methoden eine Verdrängung messen des resistenten durch den
empfindlichen Stamm messen (Abb. 22). Dieser Verdrängungsmechanismus kann
bewirken, dass bei Abwesenheit des Selektionsdruckes (sprich des Antibiotikums)
in der Umwelt die empfindlichen Varianten zu ungunsten der resistenten
gefördert werden. Entsprechende Veränderungen sind nach Umstellen des
bevorzugten Antibiotikums in Krankenhäusern beschrieben worden.
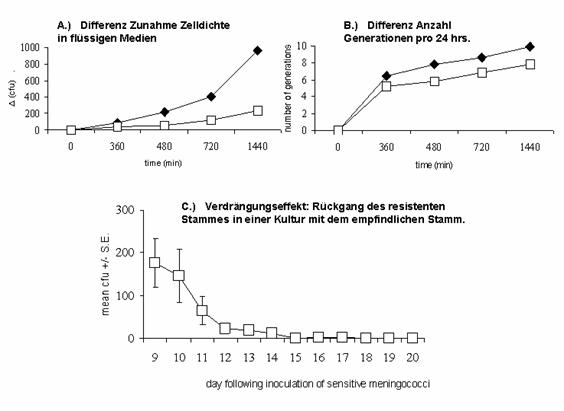
Abb. 22: Nachteil Rifampicin
resistenter N. meningitidis in
einem Milieu ohne Selektionsdruck (ohne Rifampicin).
A.) Resistente (offene Rechtecke) und empfindliche (schwarze Rauten) N. meningitidis werden getrennt in flüssigen
Kulturen angezüchtet. Nach Bestimmung der Wachstumsdichte und Normalisierung
zeigen die Kurven die Zunahme in der Zelldichte über 24 hrs
bei gleicher Ausgangssituation. B.) Umrechnung der Daten aus A. auf die Anzahl
der Generationen pro 24 hrs. C.) Ergebnisse eines
Experiments, bei dem empfindliche und resistente N. meningitidis in gleicher Dichte zum Zeitpunkt 0 in eine
flüssige Kultur eingeimpft wurden. Der Kurvenverlauf zeigt den Rückgang des
resistenten Stammes, der ab dem neunten Kulturtag (bei täglichem Umsetzen)
sichtbar wurde. Ab dem 18. Tag waren keine resistenten N. meningitidis mehr nachweisbar.